Zelluläres Räderwerk hält Proteinproduktion in Gang
 Marburg 22.12.2010 (wm/red) Drehen, schütteln und beiseite rücken – eine ausgeklügelte Choreografie sorgt in lebenden Zellen dafür, dass Aminosäuren in der vorgesehenen Reihenfolge verknüpft werden, wenn Proteine synthetisiert werden. Marburger Pharmazeuten um Professor Roland Hartmann und ihre Kooperationspartner haben jetzt mit bisher ungekannter Genauigkeit nachzeichnen können, welch komplizierte Bewegungen dabei ablaufen. Sie veröffentlichen ihre Erkenntnisse in der aktuellen Ausgabe der Wissenschaftszeitschrift „Nature“ vom zweiten Dezember 2010.
Marburg 22.12.2010 (wm/red) Drehen, schütteln und beiseite rücken – eine ausgeklügelte Choreografie sorgt in lebenden Zellen dafür, dass Aminosäuren in der vorgesehenen Reihenfolge verknüpft werden, wenn Proteine synthetisiert werden. Marburger Pharmazeuten um Professor Roland Hartmann und ihre Kooperationspartner haben jetzt mit bisher ungekannter Genauigkeit nachzeichnen können, welch komplizierte Bewegungen dabei ablaufen. Sie veröffentlichen ihre Erkenntnisse in der aktuellen Ausgabe der Wissenschaftszeitschrift „Nature“ vom zweiten Dezember 2010.
Die Biosynthese von Proteinen findet an großen Molekülkomplexen statt, den so genannten Ribosomen. Sie fungieren als Montageplattformen, an denen Ausgangsmaterialien und Werkzeuge zusammengeführt werden. Damit funktionsfähige Eiweißverbindungen entstehen, müssen die Aminosäuren genau so angeordnet werden, wie es genetisch vorgegeben ist. Forscher erklären in „Nature“ die Funktion des Ribosoms.
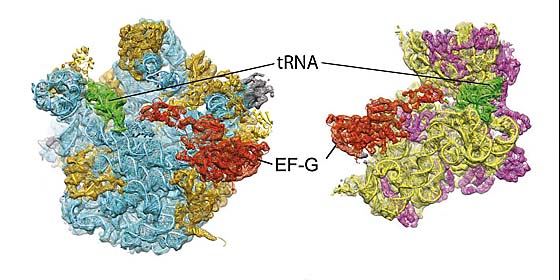
Ribosomen setzen sich aus einer großen und einer kleinen Untereinheit zusammen. Die schematische Darstellung zeigt in grün die Position der tRNA, in rot das Enzym EF-G. (Abbildung AG Hartmann, Uni Marburg)
Als Verbindungsstück zwischen der genetischen Vorlage und dem entsprechenden Protein kommt die so genannte tRNA zum Einsatz. Sie trägt jeweils eine bestimmte Aminosäure und verfügt zugleich über eine Bindungssequenz, die exakt zu definierten Stellen der Vorlage passt. Auf diese Weise gelangt jede Aminosäure an die für sie vorgesehene Position. Sobald ein tRNA-Molekül seine Aminosäure an die wachsende Kette eines Proteins abgegeben hat, wandert es entlang des Ribosoms weiter, um Platz für die nächste tRNA zu machen. Dabei bildet das Ribosom einen Komplex mit einem Enzym namens EF-G, das den Transportprozess erleichtert.
Beobachtung und Darstellung mit Elektronenmikroskop
Die beteiligten Wissenschaftler sind nun der Frage nachgegangen, wie der Ortswechsel der tRNA-Moleküle im Detail vor sich geht – ein anspruchsvolles Unterfangen, weil die Bewegung sehr schnell vor sich geht, so dass sie nur schwer zu beobachten ist. Die internationale Arbeitsgruppe rekonstruierte die aufeinander folgenden Zustände des Ribosoms, indem ein Team um Professor Christian Spahn an der Charité in Berlin die Methode der dreidimensionalen Kryo-Elektronenmikroskopie nutzte. „Bei diesem Verfahren werden die Ribosomen in flüssigem Ethan bei – 192° Celsius schockgefroren und mehrere 100.000 zweidimensionale Einzelbilder in zwei dreidimensionale Rekonstruktionen zurückprojiziert“, erläutern die Wissenschaftler.
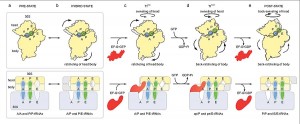
Ausgeklügelte Choreografie: Die Bewegungen bei der Proteinsynthese im Modell. Gezeigt ist die kleine Untereinheit, die Positionen der tRNAs sind farbig eingezeichnet. Die tRNAs werden durch Drehung und Verschiebung des Ribosoms von einer Position zur nächsten bewegt.
Für die tRNA lassen sich drei Positionen unterscheiden: An der ersten bindet die tRNA ans Ribosom, wobei sie eine Aminosäure trägt (A-Position); an der zweiten hat sie die Aminosäure abgegeben (P); und an der dritten verlässt die tRNA das Ribosom (E). Den Autoren ist es nun gelungen, einen neuartigen Übergangszustand zu identifizieren, bei dem sich die tRNA zwischen zwei dieser Positionen befindet. Wie die Forscher darüber hinaus feststellten, geht dieser Transport mit festgelegten Bewegungen des Ribosoms einher: Dessen beide Untereinheiten vollführen gegenläufige Drehungen und schieben dadurch die tRNA von einer Position zur nächsten. Hierbei gewährleistet das EF-G-Enzym, dass die Bewegungsrichtung beibehalten wird – wie ein Türstopper, der das Zurückschwingen verhindert.
„Unsere Einblicke in die Struktur der tRNA-Zustände legen ein Modell nahe, bei dem der tRNA-Transport durch Drehung, Einrasten und Entsperren des Ribosoms erleichtert werden“, fassen sie ihre Ergebnisse zusammen. „Diese Befunde erlauben unmittelbare strukturelle und mechanistische Einsicht in die Zwischenprodukte, die an der universell konservierten Translokation beteilgt sind.“
An der Publikation in „Nature“ sind neben Roland Hartmann und seinem Mitarbeiter Andreas Ratje weitere Wissenschaftler aus Berlin, München, Frankfurt und den USA beteiligt. Hartmanns Arbeitsgruppe am Institut für Pharmazeutische Chemie der Philipps-Universität beschäftigt sich vor allem mit der Struktur und Funktion katalytischer und regulatorischer RNA-Moleküle.
Originalpublikation Andreas H. Ratje & al.: Head swivel on the ribosome facilitates translocation by means of intra-subunit tRNA hybrid sites. Nature 468 (2 Dezember 2010), 713–716, DOI: 10.1038/nature09547










